Acidi nucleici
Acidi nucleici
I riassunti, le citazioni e i testi contenuti in questa pagina sono utilizzati per sole finalità illustrative didattiche e scientifiche e vengono forniti gratuitamente agli utenti.
GLI ACIDI NUCLEICI
Tutti gli organismi contengono acidi nucleici sotto forma di acido deossiribonucleico (DNA) e ribonucleico (RNA)
Il DNA è il depositario dell’informazione genetica che viene trascritta (cioè copiata) in molecole di RNA. L’RNA contiene il codice per sintetizzare specifiche proteine.
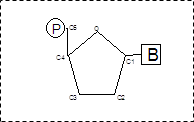 Una molecola acido nucleico è un polimero costituito da monomeri detti nucleotidi. Ciascun nucleotide è costituito da tre molecole:
Una molecola acido nucleico è un polimero costituito da monomeri detti nucleotidi. Ciascun nucleotide è costituito da tre molecole:
- uno zucchero pentoso
- una base azotata
- una molecola di acido fosforico
Uno zucchero pentoso è uno zucchero la cui molecola è costituita da 5 atomi di carbonio. La base azotata è legata al carbonio 1 dello zucchero pentoso, mentre l’acido fosforico (H3PO4) è legato al carbonio 5 che è esterno all’anello dello zucchero.
I nucleotidi si legano tra loro medianti legami fosfodiestere che collegano il C3 del pentoso di un nucleotide al C5 del nucleotide successivo. In questo modo l’acido fosforico impiega due dei suoi tre gruppi acidi nel legame fosfodiestere 3-5. il gruppo acido restante conferisce alla molecola di acido nucleico particolari caratteristiche:
- proprietà acide
- capacità di legarsi con proteine basiche (istoni)
- basofilia (la molecola può essere facilmente colorata con coloranti basici)
Lo zucchero pentoso è il ribosio nell’RNA e il deossiribosio nel DNA. La differenza tra i due è che al deossiribosio manca l’ossigeno legato al C2.
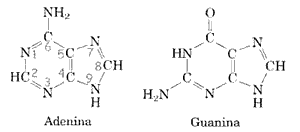
Le basi azotate si chiamano così perché contengono molti atomi di azoto e possono essere di due tipi: purine o pirimidine
 BASI AZOTATE
BASI AZOTATE


I nucleotidi che costituiscono la molecola del DNA sono:
- adenosin monofosfato
- citidin monofosfato
- guanosin monofosfato
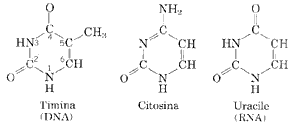
- timidin monofosfato
Quelli che costituiscono la molecola dell’RNA sono gli stessi, ad eccezione del timidin monofosfato che è sostituito dall’uridin monofosfato.
Un nucleoside è un nucleotide a cui manca il fosfato, cioè sono la combinazione di uno zucchero pentoso e di una base azotata. Per esempio, è un nucleoside l’adenosina:
Adenina → base azotata
Adenosina → nucleoside (adenina + zucchero pentoso)
Adenosin monofosfato → nucleotide (adenina + zucchero pentoso + fosfato)
L’adenosin trifosfato (ATP) è un particolare nucleotide con tre acidi fosforici, uniti tra loro con legami ad alto contenuto energetico. Infatti questa è una molecola fondamentale per la cellula perché rappresenta la principale forma di accumulo di energia.
La molecola di DNA è costituita da 2 catene polinucleotidiche le quali formano una doppia elica intorno allo stesso asse centrale. Esse sono antiparallele: i legami 3-5 fosfodiestere sono rivolti in direzione opposta. Questo vuol dire che se una catena inizia con il C3 libero e finisce con il C5 libero, l’altra catena è disposta in modo contrario.
Le due catene sono unite tra loro mediante legami a idrogeno che si instaurano tra le basi complementari. Le uniche coppie tra le quali è possibile il legame sono A-T e C-G.
Tra A e T si instaurano due legami a idrogeno mentre tra C e G se ne formano tre, quindi la coppia C-G è più stabile.
A=T C≡G
La sequenza assiale di basi lungo una catena può variare molto, ma la sequenza della catena corrispondente deve essere complementare alla prima.
Le due catene possono essere separate tra loro rompendo il legame a idrogeno tra le coppie di basi. Questo può essere fatto mediante riscaldamento o un pH alcalino (fusione o denaturazione). Dopo la denaturazione si può riottenere la conformazione a doppia elica lasciando raffreddare lentamente il DNA, in modo che le badi possano riappaiarsi (rinaturazione)
La struttura dell’RNA è simile a quella del DNA, tranne che per la presenza del ribosio invece del deossiribosio e dell’uracile invece della timina. Inoltre L’RNA è costituito da una catena singola.
Ma le molecole di RNA, avendo estese regioni complementari all’interno di una stessa catena, spesso si ripiegano e tra le basi della catena si instaurano legami a idrogeno, formando delle anse a forcina.
Ci sono tre tipi di RNA:
- RNA messaggero (mRNA): porta l’informazione genetica per la sequenza di amminoacidi
- RNA transfer (tRNA): identifica e trasporta gli amminoacidi ai ribosomi
- RNA ribosomico (rRNA): rappresenta il 50% della massa dei ribosomi

LA CROMATINA
Nelle cellule degli eucarioti il DNA non è libero ma associato a piccole proteine dette istoni, in un complesso chiamato cromatina. Esistono cinque tipi differenti di istoni, tutti di natura basica. Per questo motivo possono instaurare uno stretto legame con il DNA, di natura acida.
I quattro istoni principali, H2A, H2B, H3 e H4 hanno ciascuno una composizione simile anche nelle specie più diverse, mentre l’istone H1 è differente da specie a specie.
La cromatina può presentare vari livelli di organizzazione:
- Aspetto moniliforme: si presenta come un filo di perle, cioè come una serie di perle spesse 10 nm e collegate tra loro da un filamento di DNA. L’aspetto moniliforme non rispecchia la vera struttura della cromatina, ma è un artefatto di tecnica, in cui viene eliminato l’istone H1. Si osserva quando la cromatina viene trattata per essere studiata al microscopio elettronico
- Fibra di 10 nm: con trattamenti meno drastici è stato osservato che la cromatina si presenta come una fibra di 10 nm in cui sono presenti delle unità ripetitive a stretto contato tra loro, dette nucleosomi. In ogni nucleosoma i quattro istoni H2A, H2B, H3 e H4 sono disposti in ottameri contenenti due molecole di ciascuna proteina attorno ai quali vi si avvolge il DNA, all’esterno. I nucleosomi sono a stretto contatto tra di loro e sono localizzati a intervalli di 200 coppie di basi di DNA. L’istone H1 si trova tra un nucleosoma e l’altro. Nella fibra di 10 nm la catena di DNA da 5 a 7 volte più compatta rispetto all’aspetto moniliforme, ma ancora 1000 volte meno rispetto ai cromosomi metafasici
- Fibra spessa: ha diametro variabile tra 20 e 30 nm e rappresenta la probabile struttura della fibra inattiva. Si forma in seguito all’avvolgimento della fibra di 10 nm in un solenoide. Il DNA raggiunge una compattezza pari a 40 volte quella iniziale, e per raggiungere la compattezza di un cromosoma metafasico deve ripiegarsi ancora un centinaio di volte
I CROMOSOMI
Nel corso della divisione cellulare la cromatina si condensa a formare i cromosomi, i quali sono circa 40.000 volte più densi della fibra di 10 nm. Essi sono strutture bastoncellari che servono a poter distribuire equamente il DNA tra le cellule figlie. I componenti dei cromosomi sono 4:
- CROMATIDI alla metafase ciascun cromosoma risulta formato da due strutture simmetriche, i cromatidi, contenenti ognuna un’unica molecola di DNA. Essi sono esattamente uguali (cromatidi fratelli) e sono uniti fra loro a livello del centromero
- CENTROMERO è la regione di attacco degli elementi del fuso mitotico sul cromosoma. È situato in un tratto più sottile del cromosoma detto costrizione primaria
- TELOMERO estremità dei cromosomi contenenti l’inizio e la fine della molecola di DNA che costituisce il cromatidio
- ORGANIZZATORE NUCLEOLARE si trovano in alcuni cromosomi che presentano costrizioni secondarie in cui ci sono regioni contenenti i geni che inducono la formazione del nucleolo
TEORIA UNINEMICA: ciascun cromatidio rappresenta un’unica molecola lineare di DNA con le proteine ad essa associate
I cromosomi vengono classificati in quattro tipi a seconda della forma determinata dalla posizione del centromero:
- METACENTRICI il centromero è posto a metà dei cromatidi e quindi le braccia sono uguali
- SUBMETACENTRICI le braccia sono di lunghezza differente
- ACROCENTRICI presentano un braccio cortissimo dove si trova la costrizione secondaria all’estremità della quale c’è una regione sferoidale detta satellite
- TELOCENTRICI il centromero è posto ad un estremo
La cromatina si condensa nel corso della mitosi e si decondensa alla telofase. Ma ci sono alcune regioni dei cromosomi che non si decondensano e rimangono compatte anche durante l’interfase. Queste regioni vengono chiamate eterocromatine, mentre le altre eucromatina.
Nell’eterocramatina il DNA è molto denso e si presenta in forma di fibra di 20-30 nm cioè si tratta di DNA inattivo.
L’esempio più noto è quello della coppia di cromosomi X nelle femmine dei mammiferi: uno dei due cromosomi è attivo e rimane eucromatico mentre l’altro è inattivo e costituisce il corpo di Barr nell’interfase.
Un altro esempio è quello del gatto di Spagna. Questo esemplare presenta un mantello a macchie nere e arancioni (a placche di tartaruga), ma solo le femmine. Questo perché la macchiatura un gene contenuto nel cromosoma X diventa eterocromatico e non attivo in alcuni gruppi di cellule e non in altri. In stadi precoci dell’embriogenesi, in ogni cellula della femmina di mammifero uno dei cromosomi X diventa inattivo a caso, di conseguenza nell’adulto si forma un mosaico in cui il 50% di cellule hanno un cromosoma X attivo di origine paterna e l’altro 50% un cromosoma X attivo di origine materna
IL CICLO CELLUARE
Una cellula in accrescimento presenta un ciclo cellulare che consiste essenzialmente in due periodi: l’interfase e la divisione. L’interfase presenta tre periodi che vengono chiamati fase G1,S e G2.
- Fase G1: la cellula raddoppia le sue dimensioni e aumentano di numero organuli e molecole organiche
- Fase S: avviene la duplicazione del DNA
- Fase G2: vengono prodotte le strutture necessarie alla divisione cellulare, la cromatina inizia a spiralizzarsi
- Mitosi: avvine la divisione cellulare
Duplicazione del DNA
La duplicazione del DNA avviene nella fase S (sintesi) del ciclo cellulare d’ogni cellula.
La replicazione del DNA è semi-conservativa: questo significa che ogni molecola figlia contiene una catena parentale e una neo sintetizzata.
Il primo passo verso la replicazione semi-conservativa è la separazione delle due catene complementari nel punto in cui deve cominciare la replicazione; in modo che ciascuna catena sia libera di fungere da stampo per la polimerizzazione di una nuova catena complementare. La polimerizzazione è catalizzata dall’enzima DNApolimerasi; che seleziona i deossinucleotidi trifosfati (d-ATP, d-TTP, d-GTP, d-CTP) e li lega uno dopo l’altro con legame fosfodiestere 3’-5’. L’enzima lega il fosfato presente su un deossinucleotide trifosfato al gruppo OH legato al carbonio 3’ del deossiribosio appartenente al nucleotide terminale della catena in crescita. Nella reazione i due gruppi fosfati terminali si staccano sotto forma di pirofosfato.
La DNApolimerasi catalizza solo l’allungamento unidirezionale di una catena, cioè può aggiungere nucleotidi solo all’estremità 3’ ma non all’estremità 5’. La DNApolimerasi può catalizzare l’aggiunta di nucleotidi all’estremità 3’ di una catena esistente ma non è in grado di iniziare una nuova catena. Per iniziarla è, infatti, necessario un innesco o primer cui aggiungere (all’estremità 3’) i nucleotidi in sequenza. Nella cellula fa da innesco una breve catena di RNA. L’innesco è sintetizzato come segue:
1. prima avviene una dissociazione circoscritta delle due catene su un tratto interno alla molecola di DNA
2. un enzima detto primasi catalizza la formazione di una breve catena di RNA a partire da ribonucleotidi trifosfati (ATP, UTP, CTP, GTP) [la primasi a differenza della DNApolimerasi è capace di dare inizio ad una catena]
3. l’innesco di RNA resta appaiato allo stampo, poi arriva la DNApolimerasi che estende la catena per aggiunta di deossinucleotidi all’innesco
4. naturalmente si forma un innesco su ciascuna catena parentale e i due inneschi hanno polarità opposta e quindi le due catene figlie crescono in direzioni opposte
5. man mano che le catene figlie si estendono alla loro estremità 3’ la doppia elica si apre a cerniera in entrambe le direzioni
6. il punto di rotolamento dell’elica si chiama forcella replicativi
7. man mano che la forcella avanza lascia dietro di se una zona a singolo filamento su ciascuna delle catene parentali. A questo punto si formano nuovi inneschi che sono poi prolungati per riempire a ritroso le parti non replicate delle catene parentali
8. gli inneschi sono poi rimossi quando i frammenti sintetizzati indietro (Okazaki) incontrano l’estremità 5’ dell’innesco del tratto precedente. Quando avviene l’incontro la DNApolimerasi stacca uno dopo l’altro i nucleotidi dell’innesco e aggiunge simultaneamente deossinucleotidi
9. quando tutto l’innesco è stato rimosso le due estremità sono unite dalla DNAligasi
Nella replicazione del DNA bisogna considerare il problema che la separazione delle due catene richiede la despiralizzazione della doppia elica. La doppia elica fa un giro completo ogni 10 paia di basi. Quindi ogni 10 basi separate e svolte a livello della forcella replicativa, la doppia elica a valle deve fare un giro in direzione opposta.
In E.coli la forcella si apre alla V=60000 paia di basi al minuto, il che richiederebbe che la doppia elica a valle facesse 6000 giri al minuto (negli eucarioti la V è 10 volte inferiore). La rotazione di tutto il DNA a valle della forcella è bloccato dalle numerose proteine associate perciò in mancanza di rotazione, la torsione della doppia elica a valle ostacolerebbe la despiralizzazione e impedirebbe la replicazione. Il problema è stato risolto grazie all’enzima topoisomerasi che crea rotture transitorie in un singolo filamento a breve distanza dalla forcella replicativa. Queste rotture si creano e si riparano rapidamente ad opera della stessa topoisomerasi.
Fonte: http://riappunti.net/biologia/ACIDI%20NUCLEICI.doc
Autore del testo: non indicato nel documento di origine
Acidi nucleici
Nucleotidi e acidi nucleici
Gli acidi nucleici, DNA e RNA sono polimeri lineari di nucleotidi. Consistono
di tre componenti:
1. Uno zucchero con 5 atomi di carbono (pentoso):
- Deossiribosio, con un atomo di idrogeno legato al cabonio #2 (designato 2’)
- Ribosio, che ha un gruppo idrossilico in quella posizione
I nucleotidi che contengono deossiribosio sono i deossiribonucleotidi e sono i monomeri del DNA
I nucleotidi che contengono ribosio, ribonucleotidi, sono i monomeri del DNA
2. Una struttura ad anello contenente azoto, chiamata base. La base è legata all’atomo di carbonio 1’ del pentoso.
Nel DNA si trovano quattro basi:
- Due purine: adenina (A) e guanina (G)
- Due pirimidine: timina (T) e citosina (C)
Nel RNA si trovano le seguenti basi:
- Le stesse purine: adenina (A) e guanina (G)
- La stessa pirimidina citosina (C), ma, invece della timina, utilizza la pirimidina uracile (U)
La combinazione base + pentoso viene chiamata nucleoside
3. Uno, due o tre gruppi fosfato. Questi sono legati all’atomo di carbonio 5’ del pentoso.
Sia il DNA che l’RNA vengono sintetizzati a partire da nucleosidi trifosfati:
Per il DNA: dATP, dCTP, dGTP e dTTP
Per il RNA: ATP, CTP, GTP e UTP.
In entrambi i casi, dal momento in cui ogni nucleoside viene legato, i secondo e terzo gruppi fosfato vengono rimossi (idrolizzati).
I nucleosidi e i loro mono-, di- e tri-fosfati:
|
Base |
Nucleoside |
Nucleotidi |
||
DNA |
Adenina (A) |
Deossiadenosina |
dAMP |
dADP |
dATP |
Guanina (G) |
Deossiguanosina |
dGMP |
dGDP |
dGTP |
|
Citosina (C) |
Deossicitidina |
dCMP |
dCDP |
dCTP |
|
Timina (T) |
Deossitimidina |
dTMP |
dTDP |
dTTP |
|
RNA |
Adenina (A) |
Adenosina |
AMP |
ADP |
ATP |
Guanina (G) |
Guanosina |
GMP |
GDP |
GTP |
|
Citosina (C) |
Citosina |
CMP |
CDP |
CTP |
|
Uracile (U) |
Uridina |
UMP |
UDP |
UTP |
|
Fonte: http://www-3.unipv.it/webbio/anatcomp/freitas/2010-2011/Nucleotidi%20e%20acidi%20nucleici.doc
Autore del testo: non indicato nel documento di origine
Gli acidi nucleici
Gli acidi nucleici sono macromolecole polimeriche composti da unità chiamate nucleotidi che, a loro volta, sono costituiti da una base azotata, da un pentoso e da un gruppo fosfato. Oltre ad essere costituenti del DNA e dell'RNA i nucleotidi sono composti ricchi di energia e li troviamo anche come trasduttori secondari di segnali chimici portati da alcuni ormoni e come componenti della struttura di importanti coenzimi. (cAMP, NAD, FAD, CoA)
Le basi azotate contenute nel DNA sono rappresentate da due purine, adenina (A) e guanina (G) e da due pirimidine, timina (T) e citosina (C). Nell'RNA la timina è sostituita da una base analoga, l'uracile.
(osservare la numerazione degli atomi negli anelli. Saranno importanti perché nei nucleotidi le purine formano un legame con l'atomo di carbonio 1 del pentoso con l'N in posizione ‘9 e le pirimidine con l'N in posizione ‘1.)
|
|
purine |
pirimidine |
I pentosi dei nucleotidi sono il deossiribosio nel DNA e il ribosio nell'RNA:
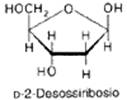
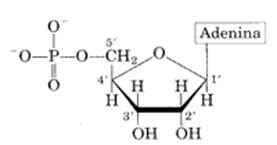
Le basi azotate si legano all'atomo di carbonio in 1' con un legame N-glicosilico (condensazione con perdita di acqua). Come detto le purine lo fanno con l'N in 9' mentre le pirimidine con l'N in 1'. Questo legame determina la formazione dei nucleosidi che prendono il nome di adenosina, guanosina, timidina, citidina e uridina. Un nucleotide è un nucleoside in cui il C in 5' del pentoso lega con legame estere, un gruppo fosfato (nucleotide monofosfato). Questa è la struttura finale dei nucleotidi presenti nel DNA e nell'RNA.
Nella figura sotto è rappresentata, come esempio, l'adenosina 5' monofosfato.
La figura sotto mostra un nucleotide trifosfato, l'ATP, a sinistra, e l'AMP ciclico (cAMP), a destra.
I nucleotidi trifosfato sono i precursori attivati nella sintesi di DNA e RNA e molti di loro, come l'ATP, sono importanti molecole che trasportano l'energia chimica. Inoltre i nucleotidi adeninici sono costituenti di molti coenzimi come il FAD, il NAD+ e il CoA.
Quando le cellule rispondono al messaggio portato da un ormone, lo fanno spesso producendo un messaggero secondario intracellulare. Uno dei più importanti è un nucleotide chiamato AMP ciclico. (in figura)
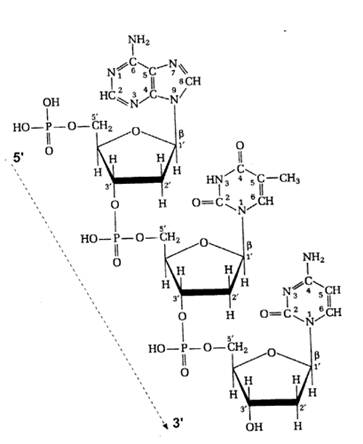
I nucleotidi del DNA e dell'RNA sono uniti tra loro mediante legami covalenti fosfodiesterici in cui il gruppo ossidrile in 5' di un nucleotide è unito al gruppo ossidrile del successivo. L'alternanza dei residui fosfato e dei pentosi formano lo scheletro degli acidi nucleici. Come si può vedere nella figura qui sotto.
E' interessante osservare che lo scheletro degli acidi nucleici ( pentoso + fosfato) è idrofilo poiché i gruppi OH dei pentosi formano legami idrogeno con l'acqua e anche purché i gruppi fosforici sono completamente ionizzati ed inoltre le loro cariche negative vengono neutralizzate da interazioni con con cariche positive di residui aminoacidici o ioni metallici.
La catena di un acido nucleico ha una polarità che ne determina il senso di crescita. Come si vede all'estremità superiore c'è un gruppo 5'P libero, mentre all'estremità inferiore c'è una estremità 3'OH libera. La crescita (sintesi) della catena procede quindi con aggiunte sul 3'OH di un nuovo nucleotide col fosfato in posizione 5'.
Le basi azotate sono idrofobiche e si disporranno in modo da evitare il contatto con l'acqua. Gli anelli eterociclici tenderanno a disporsi in pile in cui le basi risultano su piani paralleli, l'una sopra all'altra.
La doppia elica è possibile solo per interazioni ad idrogeno tra coppie di basi specifiche in modo esclusivo. Come dimostrato da Watson e Crick i legami idrogeno tra le basi sono possibili esclusivamente tra adenina e timina (2 legami) e tra guanina e citosina (3 legami).
Il DNA: la struttura
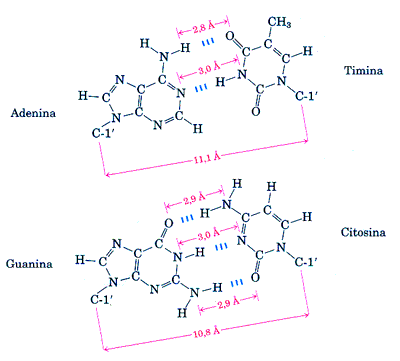
Come dimostrato da Watson e Crik la doppia elica di DNA è caratterizzata da appaiamenti delle basi azotate uniche in modo che l'adenina si leghi alla timina con due legami idrogeno e la guanina alla citosina con tre legami idrogeno. Nella figura i legami idrogeno sono evidenziati dal tratteggio.
Come si può vedere dalle distanze delle estremità delle coppie, questi appaiamenti garantiscono l'assenza di distorsioni topologiche nella struttura della molecola.
I legami così esclusivi sono alla base della tipica associazione complementare tra due catene di acido nucleico che portano alla struttura della doppia elica del DNA
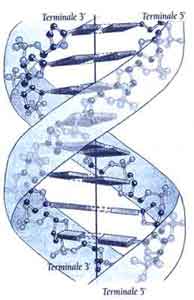
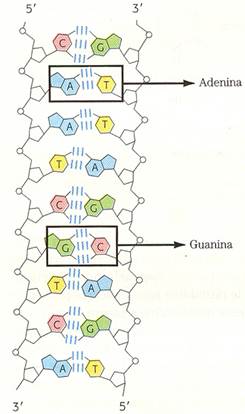
Il DNA è formato, nel suo scheletro esterno, da due catene idrofile costituite dall'alternanza di deossiribosio e fosfato che "proteggono", avvolgendole, le basi azotate, a loro volta legate al deossiribosio in C 1'. Le eliche si avvolgono attorno ad un ipotetico asse centrale (vedi figura a lato) in senso destrorso e all'interno le basi azotate, idrofobe, formano, con i loro anelli rigidamente planari, pile parallele in ogni elica che si legano con legami idrogeno alle basi complementari dell'altra elica.
La composizione in basi del DNA varia da specie a specie ma non in tessuti diversi della stessa specie e non si modifica con l'età. In tutte le specie, ovviamente, il numero delle adenina è uguale a quello delle timina così come quello delle citosina è uguale a quello delle guanina. Le eliche sono complementari l'una all'altra.
Si dice perciò che esse sono antiparallele ed il senso è chiaro se si considera che la complementarietà comporta che se un elica inizia con una terminazione 5'P, l'altra inizia con una terminazione 3'OH e viceversa. Lo stesso vale per il fine elica.
In conclusione un elica, in base ai legami fosfodiesterici, scorre in direzione 5'→ 3' e l'altra in direzione 3'→ 5'.
Geni e cromosomi
Il gene è un tratto di DNA che contiene le informazioni che possono essere decodificate al fine di produrre una catena polipeptidica. In questa definizione è racchiuso il cosiddetto "dogma centrale della biologia" i cui passaggi saranno studiati in dettaglio nella pagina dedicata al flusso dell'informazione genetica.
Le molecole di DNA, le più grandi macromolecole cellulari (una sola molecola può essere 5000 volte superiore al diametro della cellula che la contiene) sono organizzate in strutture chiamate cromosomi, contenuti e racchiuse nel nucleo delle cellule eucariotiche. Nelle cellule batteriche, l'unico cromosoma è sparso nel citoplasma. I batteri contengono anche una o molte molecole di DNA circolare extra-cromosomico chiamate plasmidi, con caratteristiche duplicative autonome. L'insieme di tutto il DNA di una cellula si chiama genoma. I virus possono contenere, come materiale genetico, o DNA o RNA. La maggioranza dei virus che infettano le piante e alcuni batteriofagi contengono una piccola (relativamente) molecola di RNA. I batteri contengono un cromosoma costituito quasi esclusivamente da geni e sequenze regolatorie. I cromosomi degli eucarioti sono molto più complessi e contengono anche brevi sequenze (10 coppie di basi) ripetute milioni di volte chiamate sequenze altamente ripetitive detto anche DNA a sequenze semplici. Questo DNA, chiamato DNA satellite, è associato, negli eucarioti a due importanti strutture: il centromero e i telomeri. Vi sono poi sequenze più lunghe (un centinaio di basi) ripetute migliaia di volte chiamate sequenze moderatamente ripetitive. Questi frammenti di DNA potrebbero rappresentare la traccia di vie evoluzionistiche abbandonate ed è quasi certo che, almeno in parte, siano un DNA di scarto.
Mentre nella maggioranza dei cromosomi batterici i geni hanno una perfetta colinearità tra sequenze nucleotidiche e sequenze aminoacidiche, negli eucarioti le sequenze nucleotidiche codificanti, esoni, sono intercalate da sequenze non traducibili, gli introni.
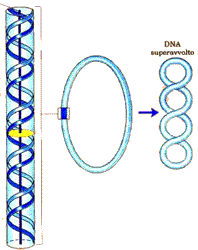
Come detto la molecola del DNA è enormemente più lunga rispetto alle dimensioni della cellula che la contiene per cui può trovare il suo spazio solo se viene compattato in una caratteristica complessità strutturale chiamata superavvolgimento. Il DNA superavvolto ha una ovvia tensione strutturale a causa dei ripiegamenti della molecola. Quando questi ripiegamenti non vi sono si dice che il DNA è in uno stato rilassato. Durante la duplicazione o la trascrizione del DNA il DNA dovrà trovarsi in uno stato di parziale rilassamento. Vi sono alcuni enzimi, le 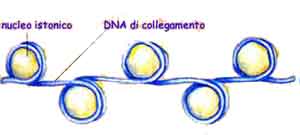 topoisomerasi, che si occupano di variare il grado di avvolgimento del DNA regolando i processi di impacchettamento o di rilassamento.
topoisomerasi, che si occupano di variare il grado di avvolgimento del DNA regolando i processi di impacchettamento o di rilassamento.
La cromatina è costituita, nella sua complessità, da parti pressoché uguali di DNA e proteine oltre che da una piccola quantità di RNA. Il DNA, in particolare, si condensa e si ordina interagendo con un gruppo di proteine basiche chiamate istoni in unità strutturali chiamate nucleosomi. Le proteine non istoniche regolano l'espressione genica.
Il flusso dell'informazione genetica
Il dogma centrale della biologia
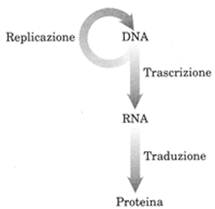 Il dogma centrale della biologia definisce ed evidenzia i processi fondamentali tramite i quali l'informazione genetica, contenuta nel nucleo nella molecola di DNA, capace di autoreplicazione, si trasferisce al citoplasma. I geni del DNA vengono, nel nucleo, trascritti in una molecola di RNA messaggero, che, passando attraverso i pori della membrana nucleare, va nel citoplasma dove, a livello dei ribosomi, viene operata, tramite il codice genetico, la traduzione dal linguaggio dei nucleotidi a quello degli aminoacidi. Le proteine così sintetizzate sono i prodotti biologici, sui quali viene trasferita l'informazione genetica che così si rende "visibile" nella loro funzionalità.
Il dogma centrale della biologia definisce ed evidenzia i processi fondamentali tramite i quali l'informazione genetica, contenuta nel nucleo nella molecola di DNA, capace di autoreplicazione, si trasferisce al citoplasma. I geni del DNA vengono, nel nucleo, trascritti in una molecola di RNA messaggero, che, passando attraverso i pori della membrana nucleare, va nel citoplasma dove, a livello dei ribosomi, viene operata, tramite il codice genetico, la traduzione dal linguaggio dei nucleotidi a quello degli aminoacidi. Le proteine così sintetizzate sono i prodotti biologici, sui quali viene trasferita l'informazione genetica che così si rende "visibile" nella loro funzionalità.
Perché l'informazione possa essere trasmessa alla generazione successiva. il DNA viene ricopiato con elevatissima precisione eliminando eventuali errori attraverso un efficace meccanismo di riparo.
La duplicazione del DNA
Conservazione e trasmissione dell'informazione alle generazioni successive
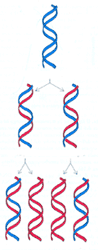
Una peculiarità della duplicazione della molecola del DNA è che essa è semiconservativa come fu dimostrato da Meselson e Sthal nel 1957. Coltivando per molto tempo cellule in un terreno contenente solo azoto pesante (15N) e centrifugandone il DNA in un gradiente di densità di CsCl, osservarono un'unica banda (a) corrispondente all'azoto pesante. Le cellule furono poi trasferite in un terreno contenente solo azoto leggero (14N). Dopo la replicazione misurarono la densità del DNA trovando una banda di sedimentazione (b) ad un livello più alto (meno denso), risultato interpretato con la formazione di un ibrido (14N/15N). Continuando la replicazione per una generazione si producevano (c) due DNA ibridi e due DNA leggeri.
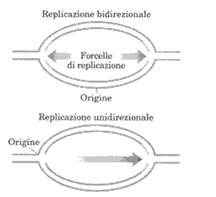
La sintesi del DNA inizia in un punto in cui la molecola viene preventivamente srotolata chiamato origine. Si forma così un'ansa di replicazione alla cui estremità troviamo le "forcelle di replicazione" e la sintesi può proseguire sia in una che in due direzione. Nel DNA batterico, circolare, le forcelle si incontrano in un punto della circonferenza opposto all'origine.
La sintesi del DNA è catalizzata da un gruppo di enzimi chiamati DNA polimerasi che possono sintetizzare solo se il punto d'allungamento è dato da una termin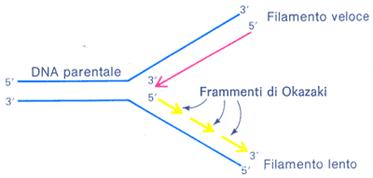 azione 3'OH libera con direzione di crescita è 5'→ 3'. Poiché le eliche sono antiparallele, ne consegue che un'elica, chiamata catena leader, o stampo, viene letta e copiata nella direzione di avanzamento della forcella e procede in modo continuo e veloce. L'altra elica dovrà essere sintetizzata con ritardo e in modo discontinuo dovendo attendere lo srotolamento dell'elica veloce. Okazaki, biologo giapponese, scoprì che la catena discontinua viene sintetizzata, con frammenti, in direzione opposta all'avanzamento della forcella.
azione 3'OH libera con direzione di crescita è 5'→ 3'. Poiché le eliche sono antiparallele, ne consegue che un'elica, chiamata catena leader, o stampo, viene letta e copiata nella direzione di avanzamento della forcella e procede in modo continuo e veloce. L'altra elica dovrà essere sintetizzata con ritardo e in modo discontinuo dovendo attendere lo srotolamento dell'elica veloce. Okazaki, biologo giapponese, scoprì che la catena discontinua viene sintetizzata, con frammenti, in direzione opposta all'avanzamento della forcella.
E' da tenere presente che ogni frammento di Okazaki termina con un'estremità 3'OH libera
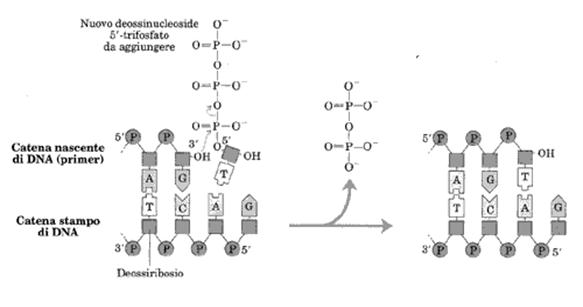
Le DNA polimerasi catalizzano la reazione di addizione di un nucleotide ad una sequenza primer e necessita di uno stampo.
Come si vede in figura sotto, l'addizione di un nucleotide trifosfato avviene in direzione 5'→ 3'. Un fosfato legato al deossiribosio in posizione 5' del nuovo nucleotide si lega con legame fosfodiesterico al deossiribosio con terminazione libera 3'OH del primer. (nella figura il deossiribosio è rappresentato da quadratini). La reazione finale addiziona un nucleotide monofosfato liberando uno ione pirofosfato (PPi).
In conclusione le DNA polimerasi necessitano di uno stampo rappresentato dall'elica antiparallela per il corretto appaiamento delle basi e di un primer, molecola di innesco che fornisca una estremità 3'OH libera. Le DNA polimerasi possono aggiungere nucleotidi ad una catena preesistente, ma non cominciare una catena ex-novo. Spesso, l'innesco è un oligonucleotide di RNA potendo la RNA polimerasi cominciare a sintetizzare una catena ex-novo. In seguito questo breve tratto di RNA sarà rimosso da una RNAasi e sostituito da DNA.
E' rilevante osservare che tutte le DNA polimerasi hanno anche un'attività 3'→ 5' nucleasica capace di rimuovere eventuali appaiamenti sbagliati. Questa attività. chiamata proofreading, lettura delle bozze, rende molto precisa ed accurata la replicazione aumentata anche da altri sistemi enzimatici di riparo.
la reazione globale catalizzata dalle DNA polimerasi è: (dNMP)n + dNTP ———→ (dNMP)n+1 + PPi(deossiNucleotideMonoP) con n nucleotidi + deossiNucleotideMonoP ———→
Gli RNA e la trascrizione
Descriviamo adesso il flusso dell'informazione genetica che dai geni contenuti nel DNA porta alla loro espressione "visibile", il fenotipo, rappresentato a livello molecolare dalle catene polipeptidiche. Come vedremo, tutti i geni attivi vengono dapprima trascritti da un enzima specifico, l'RNA polimerasi, in RNA messaggero che sono molecole capaci di passare attraverso i pori delle membrane nucleari e portare l'informazione contenuta nel DNA alle macchine molecolari sede della sintesi proteica: i ribosomi. In pratica il linguaggio dei nucleotidi viene tradotto, a livello dei ribosomi, nel linguaggio degli aminoacidi. Questa traduzione viene eseguita seguendo un "dizionario" che è il codice genetico,
A parte alcuni RNA con funzioni catalitiche o regolatorie, nella cellula vi sono 3 tipi di RNA che partecipano al flusso dell'informazione genetica:
L'RNA messaggero (mRNA) è il prodotto della trascrizione di un gene del DNA ed uscendo dal nucleo porta ai ribosomi la sequenza dei nucleotidi che sarà tradotta in sequenza aminoacidica.
L'RNA transfer (tRNA) legge le triplette del mRNA (codoni) e trasferisce il corrispondente aminoacido alla sequenza polipeptidica in crescita.
L'RNA ribosomiale (rRNA) che associato a proteine forma i ribosomi, gli organuli sede della sintesi proteica.
La trascrizione e le RNA polimerasi
Le RNA polimerasi presenti nelle cellule degli eucarioti sono 3: la RNA polimerasi I, la II e la III. Solo la polimerasi II presiede alla trascrizione dell'RNA messaggero mentre la polimerasi I trascrive i precursori dell'RNA ribosomiale e la polimerasi III presiedono alla sintesi dell' RNA transfer.
 La polimerasi II è un grosso enzima composto di 12 subunità e che richiede molte altre proteine, i fattori di trascrizione, per formare il complesso attivo. Rispetto alla DNA polimerasi, la RNA polimerasi contiene una subunità, la σ (sigma), che riconosce le sequenze del sito di inizio sintesi sul DNA, chiamato promotore e quindi non necessita di un innesco. Essa è in grado di cominciare una catena ex-novo. Una delle differenze più macroscopiche tra la replicazione e la trascrizione consiste nel fatto che nella trascrizione dell'RNA viene copiata una sola elica ed un solo piccolo tratto di essa, cioè un gene.
La polimerasi II è un grosso enzima composto di 12 subunità e che richiede molte altre proteine, i fattori di trascrizione, per formare il complesso attivo. Rispetto alla DNA polimerasi, la RNA polimerasi contiene una subunità, la σ (sigma), che riconosce le sequenze del sito di inizio sintesi sul DNA, chiamato promotore e quindi non necessita di un innesco. Essa è in grado di cominciare una catena ex-novo. Una delle differenze più macroscopiche tra la replicazione e la trascrizione consiste nel fatto che nella trascrizione dell'RNA viene copiata una sola elica ed un solo piccolo tratto di essa, cioè un gene.
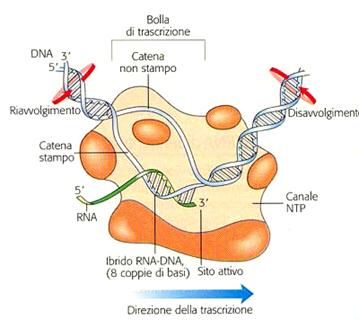
L'azione della RNA polimerasi è quella di aggiungere unità ribonucleotidiche al terminale 3'OH libero. Essa sintetizza in direzione 5'→3' avendo come stampo l'elica del DNA in direzione 3'→ 5'e quindi l'RNA messaggero che ne risulta sarà copia dell'elica non stampo che è la catena codificante. (vedi figura). In pratica l'RNA messaggero sarà una copia dell'elica 5'→3' con, l'uracile al posto della timina e il ribosio al posto del deossiribosio.
Per permettere la trascrizione, la doppia elica deve srotolarsi e aprirsi temporaneamente formando così una "bolla di trascrizione" che in figura scorre verso destra provocando lo srotolamento di altri tratti di DNA, mentre quello trascritto, a sinistra, si riavvolge. La sintesi di RNA procede fino ad un punto in cui la RNA polimerasi incontra sequenze sul DNA che favoriscono la sua dissociazione e la fine della sintesi. Il gene è stato completamente trascritto.
[La RNA polimerasi in E.Coli è un enzima di elevata complessità con un nucleo centrale costituito da cinque subunità e una sesta, chiamata σ, che si lega al nucleo dell’enzima in modo transitorio e lo dirige verso i siti di legame specifici del DNA, i siti promotori.]
Come copia la RNA polimerasi
(5’) CGCTATAGCGTTTT (3’) catena non-stampo (codificante) di DNA
(3’) GCGATATCGCAAA (5’) catena stampo di DNA
(5’) CGCUTUTGCGUUU (3’) trascritto di RNA
Modificazioni post-trascrizionali negli eucarioti
L'RNA messaggero che viene prodotto alla fine della trascrizione si chiama trascritto primario, ma in tutte le cellule eucariote questo RNA neosintetizzato subisce delle modificazioni post trascrizionali molto importanti.
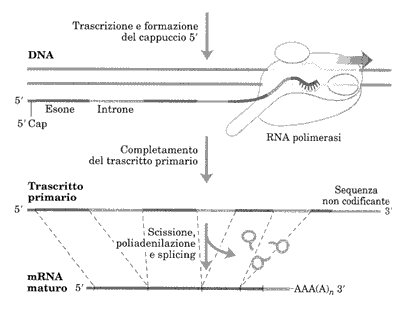
Il trascritto primario degli eucarioti contiene tutta l'informazione genetica per codificare la produzione di una catena polipeptidica ma le sequenze utili, esoni, sono intercalate da sequenze non codificanti, introni, che dovranno essere rimossi con un processo chiamato splicing. In questo modo gli esoni vengono riuniti a formare una sequenza nucleotidica contigua che specificherà per la sequenza aminoacidica funzionante.
L'RNA maturo, risultato delle modificazioni post trascrizionali del trascritto primario, degli eucarioti possiede ad un'estremità un cappuccio 5' e all'altra estremità una coda costituita da 150-200 residui di adenina: poli A. Le funzioni di queste due "appendici" non sono note del tutto anche se sappiamo che il cappuccio potrebbe partecipare al legame dell'RNA al ribosoma e che entrambe potrebbero contribuire a proteggere l'RNA dalle eventuali degradazioni enzimatiche. Tutte queste modificazioni avvengono nel nucleo, prima che l'mRNA lo lasci attraverso i pori della membrana nucleare.
Il codice genetico
La sintesi delle proteine è un meccanismo complesso le cui informazioni sono contenute nella sequenza nucleotidica del DNA e che, con la duplicazione, vengono tramandate alle generazioni successive. La trascrizione del DNA in mRNA assicura la conservazione delle informazioni contenute nei geni e la loro esportazione alle macchine molecolari che dovranno eseguire la traduzione, i ribosomi. L' RNA messaggero è dunque una catena che contiene le sequenze nucleotidiche dell'elica codificante del DNA. Ma come vengono lette tali sequenze per poi tradurle nel linguaggio degli aminoacidi? E' ovvio che non può esserci corrispondenza tra un nucleotide e un aminoacido essendo diversa e largamente insufficiente la "numerosità" dei nucleotidi.
Anche se si considerassero le doppiette, le combinazioni possibili di quattro basi, prese a due a due sarebbe 42 =16, ancora insufficiente per codificare tutti e venti gli aminoacidi.
Accurati studi hanno dimostrato che la sequenza di basi dell'mRNA viene letta a triplette. Le combinazioni possibili sarebbero così 43 = 64, più che sufficienti per codificare i venti aminoacidi. Sulla base di ingegnosi esperimenti scientifici si è potuto alla fine decifrare tutto il codice genetico e le sue proprietà. (vedi figura)
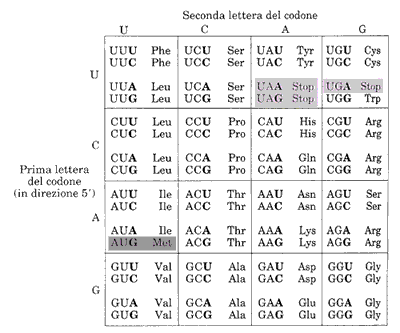
Il codice genetico è infatti composto dalle 64 triplette possibili di cui 61 codificanti per un determinato aminoacido e tre di fine lettura o codoni non senso (stop in figura). La tripletta AUG, evidenziata in figura, è il codone d'inizio della lettura sia negli eucarioti che nei procarioti. Se però questa tripletta non è all'inizio della lettura, essa codifica per l'aminoacido metionina. Tutte le triplette nell'mRNA prendono il nome di codoni.
La sequenza aminoacidica di una proteina è quindi definita dalla sequenza delle triplette del DNA portate nel citosol dalla sua "fotocopia" rappresentata dall'mRNA.
Il codice genetico non è sovrapposto il che vuol dire che nessun nucleotide fa parte contemporaneamente di più di un codone. Esso viene letto senza punteggiatura. Come è ovvio data la diversa numerosità, non esiste una corrispondenza biunivoca tra triplette e aminoacidi nel senso che ad una tripletta, delle 61, corrisponde un aminoacido ma più aminoacidi possono essere codificati da più di una tripletta. Si dice per questo che il codice è non ambiguo, poiché nessun codone specifica per più di un aminoacido e degenerato, non dando però a questo aggettivo il significato negativo che ha nell'accezione corrente. La degenerazione del codice invece è la caratteristica più sorprendente per la quale per molti aminoacidi la terza base può oscillare liberamente. La degenerazione è, in definitiva, anche un sistema di difesa dalle mutazioni puntiformi spontanee che se colpiscono la terza base possono non alterare la sequenza aminoacidica e quindi la conformazione tridimensionale e l'efficienza della proteina. Il codice è universale.
L’RNA transfer
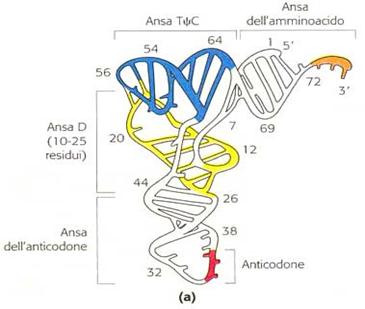
Le molecole che si incaricano di leggere i codoni e di trasportare i relativi aminoacidi sono i tRNA (RNA transfer). Sono molecole relativamente piccole a singola elica ripiegata in una precisa struttura tridimensionale. Essi sono composti da 73 a 93 nucleotidi.
Una visione bidimensionale è quella della figura sotto in cui si vedono alcune anse. All’estremità 3’ troviamo l’ansa dell’aminoacido (meglio visibile nella visione tridimensionale). Essa contiene in tutti i tRNA una tripletta CCA(3’). Essa trasporta un aminoacido legato con legame estere tra il gruppo carbossilico dell’aminoacido e quello ossidrilico del residuo di adenosina. Vedi figura alla pagina successiva.
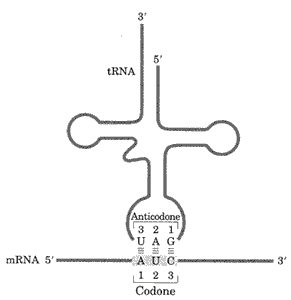
Essi sono formati da quattro anse che li fanno somigliare ad un trifoglio. Come si vede, il tRNA ha l’ ansa dell’ anticodone che legge l'mRNA in direzione 5'→ 3', per cui cominciano la lettura col nucleotide libero in 3'. L'ansa dell'aminoacido, in terminazione
3' contiene la tripletta CCA, ed è la zona in cui si lega l'aminoacido corrispondente. Le altre anse, chiamate ansa D e ansa TΨC, contengono nucleotidi inusuali come la diidrouridina (D) e la pseudouririna (Ψ) e contribuiscono alle importanti interazioni che mantengono il corretto ripiegamento del transfer.
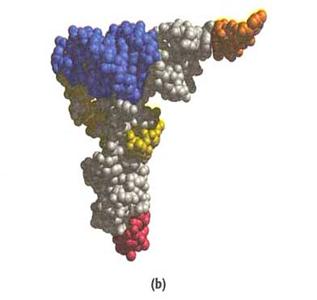
Struttura tridimensionale del tRNA.
La struttura tridimensionale mostra una forma a L rovesciata ma ci da una migliore visione delle anse dell’aminoacido e quelle dell’anticodone.
|
|
I ribosomi : sono le macchine molecolari deputate alla sintesi delle proteine. Gli studi sui ribosomi batterici hanno chiarito molto sia della loro composizione sia del loro ruolo nella sintesi proteica. Essi sono costituiti per il 65% da rRNA e per il 35% da proteine. Sono composti di due subunità diseguali con un coefficiente di sedimentazione rispettivamente di 30S per la subunità minore e di 50S per quella maggiore; le due subunità unite hanno un coefficiente di 70S mentre quelli degli eucarioti hanno una coefficiente di sedimentazione di 80S con subunità di 60S e 40S. I ribosomi possono essere liberi e sparsi nel citosol, come nei procarioti, oppure legati a quel complicato intreccio di canali e di cisterne delimitati da membrane che prende il nome di reticolo endoplasmatico rugoso per la presenza, appunto, dei ribosomi. L'associazione delle due subunità lascia una fessura attraverso la quale passa l'mRNA e lungo la quale il ribosoma si muove durante la sintesi delle catene polipeptidiche. Come si vedrà alla subunità minore si legherà l’mRNA ma il complesso d’inizio sintesi sarà composto da entrambe le subunità che presentano nel loro interno 3 siti importanti chiamato sito A (aminoacilico), sito P (peptidilico) in entrambe le subunità e sito E (sito d’uscita, exit) nella subunità maggiore, 50S.
La sintesi proteica
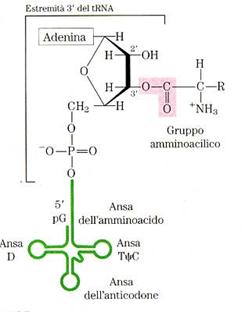
STADIO 1: attivazione degli aminoacidi
Nella prima fase della sintesi proteica, che avviene nel citoplasma, i 20 diversi aminoacidi si legano tramite legame estere tra il gruppo carbossilico dell’aminoacido e quello ossidrilico del residuo di adenosina all'estremità 3' del loro tRNA, ad opera di un enzima chiamato aminoacil-tRNA sintetasi. L'aminoacido è così attivato ed è pronto per essere trasportato sulla catena polipeptidica in crescita dove avverrà la formazione del legame peptidico con l'aminoacido successivo.
In figura si vede la struttura generale di un aminoacil-tRNA che è l’aminoacido legato al suo tRNA e attivato per la sintesi. Esiste generalmente in ogni organismo una aminoacil-tRNA sintetasi per ciascun aminoacido.
La reazione globale è endoergonica e può avvenire con la contemporanea idrolisi dell’ATP Mg++ dipendente.
STADIO 2: Formazione del complesso d’inizio.
La subunità minore 30S si ancora ai due fattori d’inizio IF1 e IF3 L’mRNA si lega alla subunità 30S.
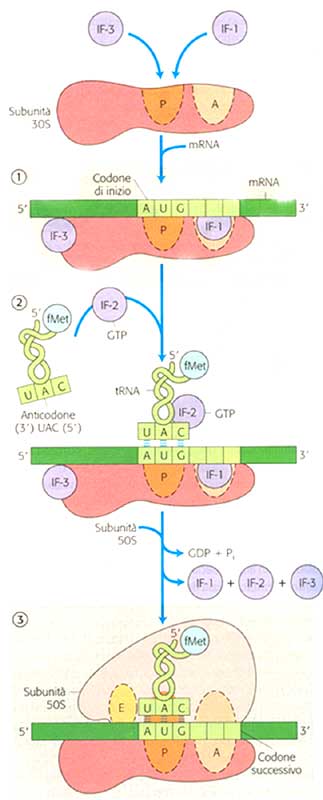
La sintesi proteica comincia con la lettura del codone d'inizio sintesi AUG che specifica per un residuo di metionina (met) ammino-terminale (formil-metionina nei batteri fMet) Il tRNA per la metionina (fMet) avrà l'anticodone UAC.
L'innesco della sintesi proteica necessita di tre proteine chiamati fattori d'inizio (IF-1, IF-2, IF-3). Seguire la figura:
(1) I ribosomi presentano nelle loro subunità due siti, il sito P (peptidilico) e sito A (aminoacilico). Nella figura il sito A è inizialmente bloccato dal IF-1 per impedirne eventuali legami.
La subunità ribosomiale minore (30S) si lega ai fattori d'inizio IF-1 e IF-3 e su di essa ha luogo il legame con l'mRNA in direzione
5'→ 3'. Sul ribosoma esiste una cosiddetta sequenza consenso (sequenza di Shine-Dalgarno) che immobilizza l'RNA ribosomiale al tRNA indirizzando il codone d'inizio AUG nella posizione giusta per l'inizio della traduzione: nel sito P. Osservare che solo il fMet-tRNA si posiziona nel sito P, tutti gli altri si posizioneranno nel sito A. Come si vede in figura i siti P ed A contengono lo "spazio" per l'alloggio di una tripletta per sito.
(2) A questo punto al sito P si addiziona il tRNA portante la metionina "attivato" dall'IF-2 legato al GTP. Si ha il corretto appaiamento dell'anticodone che scorre in direzione 3'→ 5'al codone dell'mRNA. Successivamente a questo voluminoso complesso si aggiunge la subunità 50S (3) e tutti i fattori d'inizio si staccano dal ribosoma.
Il fattore d’inizio è dunque composto dal ribosoma intero con entrambe le due subunità. Nel sito P la fMet-tRNA fa, con l’anticode del transfer, legami idrogeno con le basi complementari dell’mRNA.
STADIO 3 Allungamento della catena polipeptidica con formazione di legami peptidici
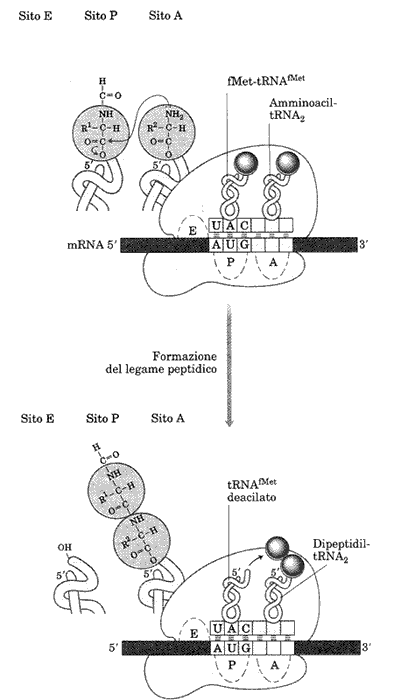
Dopo che si è insediato il primo tRNA e quindi il primo aminoacido, la metionina o la formilmetionina, si passa alla fase dell'allungamento della catena che consiste nella formazione dei legami peptidici del nuovo aminoacido con quello preesistente. Così la crescita della catena presuppone che la metionina resti il primo aminoacido (col gruppo N terminale a sinistra). Durante l'allungamento entrano in azione i fattori d'allungamento che sono tre proteine solubili nel citosol.
Il secondo aminoacil-tRNA, legato ai fattori d'allungamento, entra nel sito A del ribosoma intero. L’anticodone sarà ovviamente complementare alla tripletta (codone) del mRNA posta sul sito A.
Il legame peptidico si forma per trasferimento della formilmetionina sul nuovo aminoacido. (vedi dettaglio). Si forma il legame peptidico che impegna il gruppo carbossilico della metionina e quello amminico del 2° aminoacido. Si ottiene così un dipeptide e un tRNA "scarico", nel sito P. I legami peptidici sono catalizzati dall'enzima peptidil transferasi.
A questo punto il ribosoma si sposta lungo l'mRNA in direzione 3' di un solo codone con conseguente traslocazione del complesso portante il dipeptide sul sito P. Il sito A si libera per essere "letto" da un nuovo tRNA che trasporta l'aminoacido del nuovo codone. Come si vede nella subunità 50S è presente un altro sito, E (exit), che è il sito d'uscita dei tRNA deacilati. (privi dell’aminoacido)
La traslocazione e lo spostamento richiedono consumo d'energia.
STADIO 4 : Fine sintesi.
Il ciclo continua fino a che l'mRNA non presenta sul sito A un codone UAA, UAG o UGA, codoni di fine lettura.
Nei batteri intervengono tre fattori di rilascio che provvedono ad idrolizzare il legame terminale del peptidil-tRNA, il rilascio del polipeptide e la dissociazione delle subunità dei ribosomi, pronti così a cominciare una nuova sintesi. 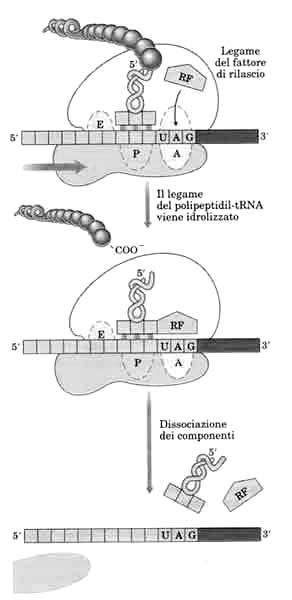 (vedi figura sotto)
(vedi figura sotto)
STADIO 5 : modificazioni post sintetiche
La catena polipeptidica neosintetizzata si libera con l'estremità N-terminale a sinistra e subisce tutte quelle modificazioni che la sua sequenza aminoacidica le consentono. Assumerà quindi la sua conformazione che permetta il massimo numero di legami idrogeno, di interazioni di van der Waals, di interazioni ioniche e idrofobiche oltre i ponti disolfuro tra cisteine.
Alcune proteine potranno raggiungere la loro conformazione biologicamente attiva solo dopo aver subito modificazioni post-traduzionali come ad esempio la rimozione della metionina iniziale.
Possono essere aggiunti alla proteina catene laterali di carboidrati formando le glicoproteine, oppure, fondamentale nelle proteine con funzione enzimatica, è l'aggiunta dei gruppi prostetici.
Fonte: http://xoomer.virgilio.it/cyrano2510/Bio-molecolare.doc
Autore del testo: non indicato nel documento di origine
Acidi nucleici
Visita la nostra pagina principale
Acidi nucleici
Termini d' uso e privacy